Zgadzam się z odpowiedzią Aleksieja Zajcewa (i omówieniem tej odpowiedzi), że standaryzacja jest dobra do zrobienia z różnych powodów również na wynikach. Chciałbym jednak tylko dodać przykład, dlaczego standaryzacja wyników może być ważna.
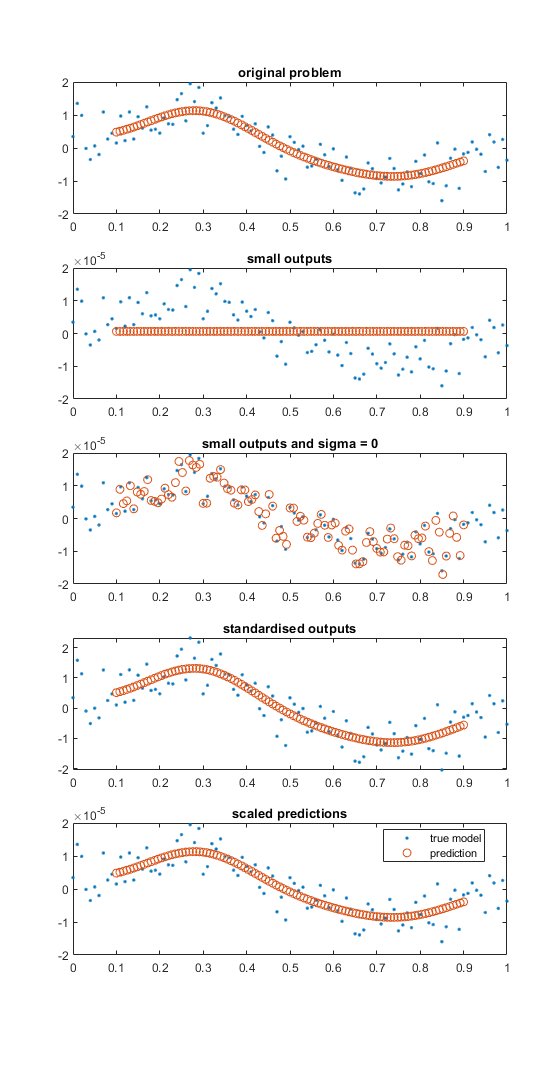
Przykładem, w którym normalizacja / standaryzacja wyjścia jest ważna, to małe i zaszumione wartości wyjściowe. Poniższy rysunek ilustruje to na bardzo prostym przykładzie. Kod MATLABa poniżej rysunku.
Niebieskie kropki reprezentują zaszumione próbki funkcji sin (). Pomarańczowe / czerwone kółka reprezentują przewidywane wartości procesu Gaussa.
- W górnym dolnym wykresie amplituda jest równa jedności (z szumem).
- W drugim wykresie pomocniczym wartości wyjściowe zostały przeskalowane za pomocą 1e-5 i widzimy, że model procesu Gaussa (z ustawieniami domyślnymi) przewiduje model stały. Domyślne ustawienia optymalizują parametr szumu i mają dość wysoką dolną granicę.
- W trzecim wykresie pomocniczym parametr szumu jest ustawiony na zero i nie jest optymalizowany. W tym przypadku model zbyt mocno pasuje.
- Czwarty wykres pomocniczy przedstawia standardowe wyniki i model dopasowany do tych danych.
- W ostatnim wykresie podrzędnym wyniki są skalowane wstecz z operacji standaryzacji (nie skalowane z powrotem do wartości pierwotnych), a także wartości przewidywane są skalowane. Zwróć uwagę, że przewidywane wartości są skalowane przy użyciu średniej i odchylenia standardowego ze standaryzacji danych uczących.
, funkcja valid_normgp ()
% małych wyników
% danych
x = 0: 0,01: 1; x = x (:);
xp = przestrzeń linowa (0,1, 0,9, długość (x)); xp = xp (:);
% modelu bez hałasu
y = sin (2 * pi * x) + 5e-1 * randn (length (x), 1);
% trenuje i przewiduje model GP
mdl = fitrgp (x, y);
yp = przewidzieć (mdl, xp);
postać
subplot (5, 1, 1)
plot (x, y, '.')
czekaj
plot (xp, yp, 'o')
tytuł ('oryginalny problem')
%% sprawia, że wyjścia są małe (poniżej dolnej granicy szumu)
ym = y / 1e5;
% trenuje i przewiduje model GP
mdlm = fitrgp (x, ym);
ypm = przewidzieć (mdlm, xp (:));
subplot (5, 1, 2)
plot (x, ym, '.')
czekaj
plot (xp, ypm, „o”)
tytuł („małe produkty”)
%% wyświetla małe i ustawia sigma = 0
% trenuje i przewiduje model GP
mdlm1 = fitrgp (x, ym, 'Sigma', 1e-12, 'ConstantSigma', true, 'SigmaLowerBound', eps);
ypm1 = przewidzieć (mdlm1, xp (:));
subplot (5, 1, 3)
plot (x, ym, '.')
czekaj
plot (xp, ypm1, 'o')
tytuł ('małe wyniki i sigma = 0')
%% normalizuj / standaryzuj
nu = średnia (ym);
sigma = std (ym);
yms = (ym - nu) / sigma;
% trenuje i przewiduje model GP
mdlms = fitrgp (x, yms);
ypms = przewidzieć (mdlms, xp (:));
subplot (5, 1, 4)
plot (x, yms, '.')
czekaj
plot (xp, ypms, „o”)
tytuł ('standardowe wyjścia')
% rescale
ypms2 = ypms * sigma + nu;
subplot (5, 1, 5)
plot (x, ym, '.')
czekaj
plot (xp, ypms2, „o”)
tytuł („skalowane prognozy”)
legenda („prawdziwy model”, „prognoza”, „lokalizacja”, „najlepsza”)
koniec