Przykład z normalnymi danymi. Załóżmy, że rzeczywiste dane to losowa próbka o rozmiarze $ n = 200 $ z $ \ mathsf {Norm} (\ mu = 100, \ sigma = 15), $ ale nie znasz $ \ mu $ lub $ \ sigma $ i postaraj się je oszacować. W poniższym przykładzie oszacowałbym wartość $ \ mu $ na podstawie $ \ bar X = 100,21 $ i $ \ sigma $ do $ S = 14,5, $ Oba szacunki są całkiem dobre. (Symulacja i obliczenia w R.)
set.seed (402) # dla odtwarzalności
x = rnorm (200, 100, 15)
średnia (x); sd (x)
# [1] 100,2051 # aprx 100
# [1] 14.5031 # aprx 15
Załóżmy teraz, że brakuje 25% tych danych. (To duża część, ale jestem
próbując wskazać punkt.) Jeśli zastąpię brakujące obserwacje średnią ze 150 niebrakujących obserwacji, zobaczmy, jakie są moje oszacowania $ \ mu $ i $ \ sigma $ będzie.
x.nonmis = x [51: 200] # dla uproszczenia załóżmy, że brakuje pierwszych 50
x.imputd = c (rep (średnia (x.nonmis), 50), x.nonmis)
długość (x.imputd); mean (x.imputd); sd (x.imputd)
# [1] 200 # „x.imputd” ma odpowiednią długość 200
# [1] 100,3445 # aprx 100
# [1] 12,58591 # znacznie mniejszy niż 15
Teraz szacujemy $ \ mu $ na $ \ bar X_ {imp} = 100,3, $ co nie jest złym oszacowaniem, ale potencjalnie (jak tutaj) gorzej niż średnia z rzeczywistych danych. Jednak obecnie szacujemy, że $ \ sigma $ jako $ S_ {imp} = 12,6, $ jest dość nieco poniżej zarówno prawdziwego $ \ sigma $ , jak i jego lepszego oszacowania 14,5 z rzeczywistych danych.
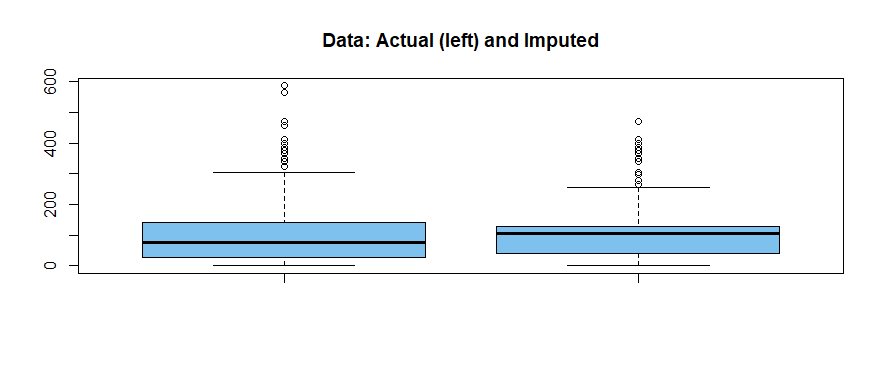
Przykład z danymi wykładniczymi. Jeśli dane są mocno przekrzywione w prawo (jak w przypadku danych z populacji wykładniczej), zastąpienie brakujących danych średnią danych bez brakujących danych może maskować skośność, tak aby możemy być zaskoczeni, że dane nie odzwierciedlają tego, jak ciężki jest prawy ogon populacji.
set.seed (2020) # dla odtwarzalności
x = rexp (200, .01)
średnia (x); sd (x)
# [1] 108.0259 # aprx 100
# [1] 110,1757 # aprx 100
x.nonmis = x [51: 200] # dla uproszczenia załóżmy, że brakuje pierwszych 50
x.imputd = c (rep (średnia (x.nonmis), 50), x.nonmis)
długość (x.imputd); mean (x.imputd); sd (x.imputd)
# [1] 200
# [1] 106,7967 # aprx 100
# [1] 89,21266 # mniejsze niż 100
boxplot (x, x.imputd, col = "skyblue2", main = "Dane: rzeczywiste (po lewej) i przypisane")
Wykres pudełkowy pokazuje więcej skośności w rzeczywistych danych (wiele obserwacji z dużym ogonem)
niż w danych „imputed”.
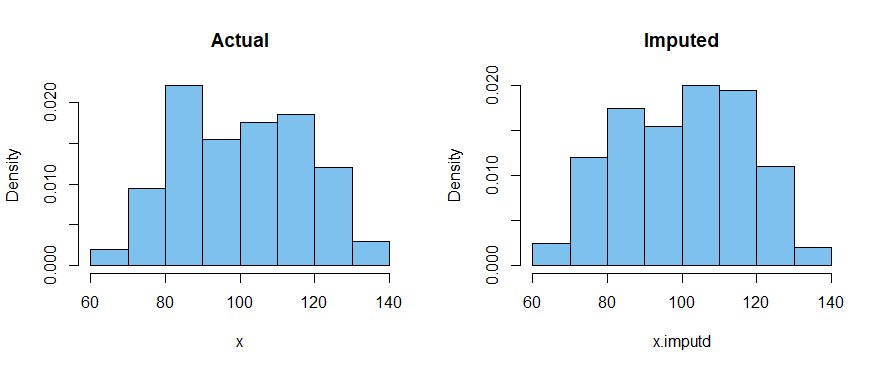
Przykład z danymi bimodalnymi. Ponownie tutaj, kiedy podstawimy brakujące wartości przez
średnia z obserwacji bez brakujących danych, odchylenie standardowe populacji jest niedoszacowane. Być może poważniej, duża liczba imputowanych wartości w środku „imputowanej” próbki maskuje bimodalny charakter danych.
set.seed (1234) # dla odtwarzalności
x1 = rnorm (100, 85, 10); x2 = rnorm (100, 115, 10)
x = próbka (c (x1, x2)) # losowa kolejność mieszania
średnia (x); sd (x)
# [1] 99.42241
# [1] 18.97779
x.nonmis = x [51: 200] # dla uproszczenia załóżmy, że brakuje pierwszych 50
x.imputd = c (rep (średnia (x.nonmis), 50), x.nonmis)
długość (x.imputd); mean (x.imputd); sd (x.imputd)
# [1] 200
# [1] 99.16315
# [1] 16.41451
par (mfrow = c (1,2))
hist (x, prob = T, col = "skyblue2", main = "Actual")
hist (x.imputd, prob = T, col = "skyblue2", main = "Imputed")
par (mfrow = c (1,1))
Ogólnie: Zastępowanie brakujących danych średnimi danymi bez brakujących danych powoduje, że SD populacji jest niedoszacowany, ale może również zaciemniać ważne
cechy populacji, z której próbkowano dane.
Uwaga: jak zauważa @ benso8, użycie średniej z danych bez brakujących danych w celu zastąpienia brakujących obserwacji jest nie zawsze złym pomysłem. Jak wspomniano w pytaniu, ta metoda ogranicza zmienność. Z pewnością każdy schemat będzie miał wady
za radzenie sobie z brakującymi danymi. Pytanie dotyczyło spekulacji na temat możliwych niedogodności innych niż redukcja wariancji dla tej metody. Próbowałem zilustrować kilka możliwości w moich dwóch ostatnich przykładach.
Wstępna metoda alternatywna: nie jestem ekspertem w eksploracji danych. Dlatego bardzo wstępnie proponuję alternatywną metodę. Nie twierdzę, że to nowy pomysł.
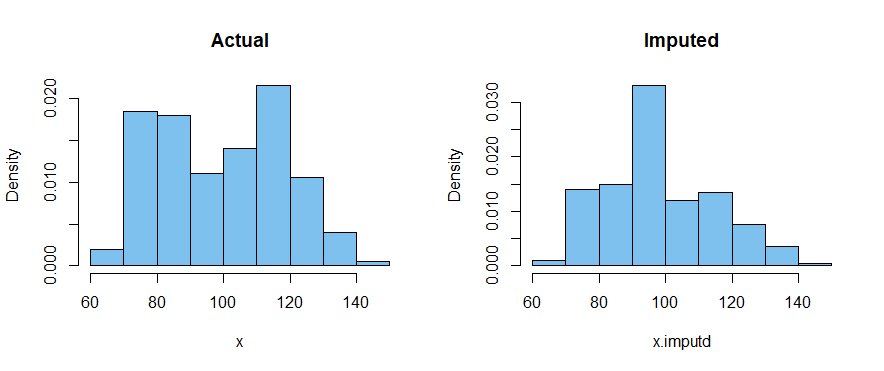
Zamiast zastępować wszystkie $ m $ brakujące elementy średnią próbną elementów, których nie brakuje, można wziąć losową próbkę o rozmiarze $ m $ spośród niebrakujących obserwacji i przeskaluj go tak, aby $ m $ elementy miały tę samą średnią i odchylenie standardowe, co niebrakujące dane . Następnie połącz przeskalowane elementy $ m $ z elementami bez brakujących danych, aby otrzymać „przypisaną” próbkę z prawie taką samą średnią i odchyleniem standardowym, co niebrakująca część próbki.
Wynik nie powinien systematycznie lekceważyć SD populacji i może lepiej zachować cechy populacji, takie jak skośność i bimodalność. (Komentarze mile widziane.)
Pomysł ten jest rozważany poniżej dla danych bimodalnych:
set.seed (4321) # dla odtwarzalności
x1 = rnorm (100, 85, 10); x2 = rnorm (100, 115, 10)
x = próbka (c (x1, x2)) # scrmble
średnia (x); sd (x)
# [1] 100,5299
# [1] 17.03368
x.nonmis = x [51: 200] # dla uproszczenia załóżmy, że brakuje pierwszych 50
an = średnia (x.nonmis); sn = sd (x.nonmis)
x.subt = sample (x.nonmis, 50) # tymczasowe nieskalowane substytuty
as = średnia (xsubt); ss = sd (x.subt)
x.sub = ((x.subt - as) / ss) * sn + an # skalowane podstawniki
x.imputd = c (x.sub, x.nonmis)
mean (x.imputd); sd (x.imputd)
# [1] 100.0694 # aprx to samo co średnia z braku braków
# [1] 16.83213 # aprx to samo SD systemu operacyjnego, co nie brakuje
par (mfrow = c (1,2))
hist (x, prob = T, col = "skyblue2", main = "Actual")
hist (x.imputd, prob = T, col = "skyblue2", main = "Imputed")
par (mfrow = c (1,1))